Descobrimos um mecanismo generalizado que descreve como uma bactéria utiliza genes adquiridos por transferência lateral de genes. Esse mecanismo ajuda na compreensão da evolução do genoma bacteriano e tem aplicações práticas em biologia sintética e biotecnologia.
No mundo animal, a reprodução sexuada mistura o material genético dos pais, aumentando a diversidade genética e a capacidade adaptativa de uma espécie. Embora as bactérias não compartilhem o mesmo mecanismo de reprodução sexuada do mundo animal, elas são capazes de adquirir sequências de DNA de uma outra bactéria ou de seu ambiente por um mecanismo conhecido por Transferência lateral de genes. Esse mecanismo permite diversificação do repertório genético de uma bactéria e aquisição de novas funções adaptativas. O processo de transferência lateral de genes tem importância fundamental na biologia básica dos microrganismos e em aplicações de engenharia genética. Esse mecanismo também é de relevância na saúde pública, pois é responsável pela alta incidência de resistência à antibióticos em hospitais.
Um questão pendente nesse processo é como uma bactéria utiliza os genes adquiridos. Não basta apenas carregar a sequência de DNA de um gene, mas é preciso que o gene seja expressado (produzido) na forma de uma proteína. A expressão gênica precisa ser bem regulada para trazer benefício adaptativo à bactéria. O gene sem ser expresso não traz uma função nova (por exemplo, resistência a antibióticos) e um gene expressado em excesso é tóxico à célula (veja a figura abaixo). A expressão gênica é controlada por sequências de DNA específicas, também conhecidas como sequências reguladores, que indicam o contexto e a quantidade em que a proteína deve ser produzida.
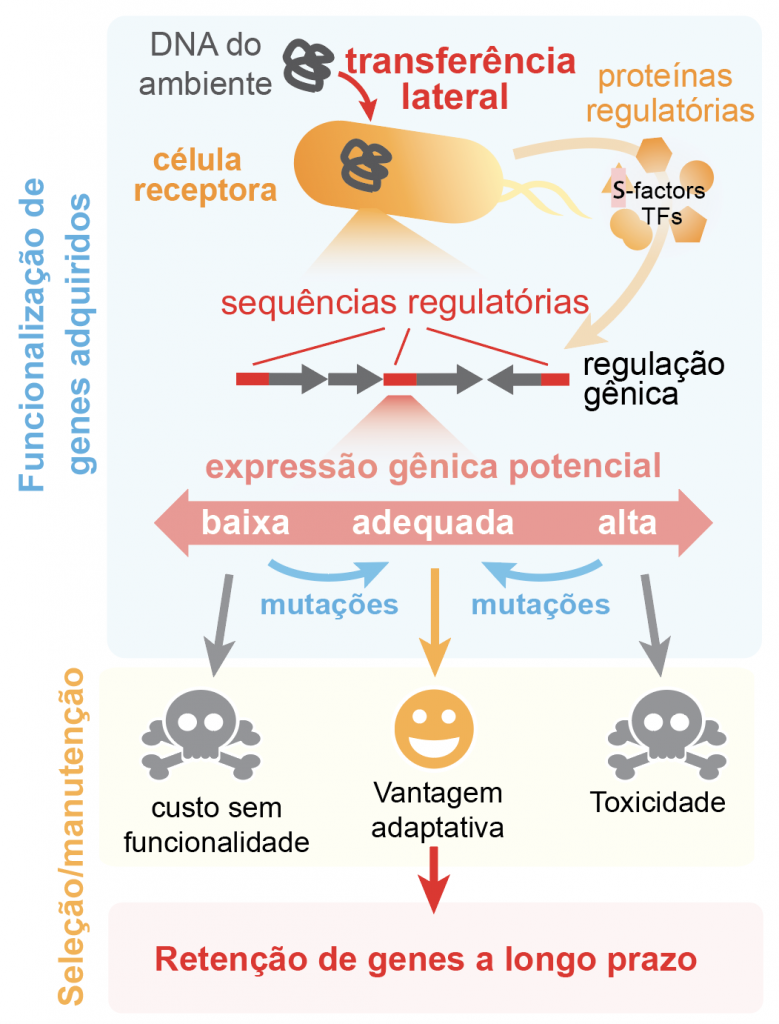
Para investigar como as sequências reguladoras de um organismos são reconhecidas por outro, nós desenvolvemos um novo método que simula a transferência lateral de sequências reguladoras experimentalmente. Cerca de 30 mil sequências regulatórias de um grupo de 184 genomas representativos da diversidade de bactérias na natureza foram avaliadas em 3 espécies de bactérias receptoras: Baccilus subtilis, Escherichia coli e Pseudomonas aeruginosa.
Os resultados foram surpreendentes. As três espécies reconheciam o mesmo padrão de sequência regulatória para expressar os genes adquiridos. Porém, de forma curiosa, a capacidade de expressão gênica entre elas foi bem distinta. B. subtilis mostrou a menor compatibilidade regulatória, expressando cerca de 29% das sequências testadas, seguida por E. coli (61%) e P. aeruginosa (83%). Por fim, descobrimos o motivo da diferença de capacidade de expressão gênica. Organismos com maior frequência de nucleotídeos GC no genoma (no caso, P. aeruginosa) expressam genes mais facilmente do que organismos de menor frequência GC (no caso, B. subtilis). Nós propusemos um modelo matemático que permite prever e quantificar a magnitude da expressão gênica.
A descoberta do nosso estudo é de interessante amplo em biologia básica e aplicada. Esse modelo pode ajudar a entender porque algumas construções utilizadas em biotecnologia funcionam em uns organismos, mas não em outros. A capacidade quantitativa do modelo pode ser utilizada para calibrar o sinal de expressão gênica de acordo com o organismo de interesse e a aplicação prática. Finalmente, a compatibilidade regulatória pode ter um papel para entender a manutenção de genes adquiridos lateralmente e justifica a observação de genomas largos serem mais comuns em organismos de alto conteúdo GC.
por Antonio L. C. Gomes
…………………………………………………………
Referências
1. Gomes, A. L. C. et al. Genome and sequence determinants governing the expression of horizontally acquired DNA in bacteria. The ISME Journal, doi:10.1038/s41396-020-0696-1 (2020).
2. Johns, N. I. et al. Metagenomic mining of regulatory elements enables programmable species-selective gene expression. Nature Methods 15, 323-329, doi:10.1038/nmeth.4633 (2018).
Imagem de capa de Gerd_Altmann, por Pixabay.
